
%matplotlib inline
import numpy as np
A partir de una lista de Python:
lista = [1, 2, 3, 4 , 5]
type(lista)
a = np.array(lista)
a
type(a)
Podemos conocer el tipo de datos a través de dtype:
a.dtype
Incluso podemos definir el tipo de dato al momento de la creación de arreglo:
a_complejo = np.array(lista, dtype=complex)
a_complejo
a_complejo.dtype
o cambiar el tipo de dato:
a_no_mas_complejo = a_complejo.astype(np.int64)
a_no_mas_complejo
Si queremos ver algunas características del arreglo a:
a.ndim # dimensión
a.shape # 5 x 1
len(a) # elementos en la primera dimension
b = np.array([[0, 1, 2], [3, 4, 5]]) # arreglo 2 x 3
b
b.ndim
b.shape
len(b) # elementos en la primera dimensión
c = np.array([[[1], [2]], [[3], [4]]])
c
c.shape
a = np.arange(10) # otra forma de generar un arreglo: a través de funciones específicas de numpy
a
a[0], a[2], a[-1]
mis_indices = [0, 2, -1] # puedo listar índices
a[mis_indices] # y luego indexar por esa lista ("fancy indexing")
a
a[2:9] # corta en intervalos
a[2:9:3] # puedo especificar cada cuánto cortar
a[::2]
a[3::2]
a[-2:]
Un esquema siempre ayuda...

Si copiamos una lista en Python... si modificamos la copia, no se modifica la lista originalmente copiada:
%%python
# python cell magic, lindo... no?
a = [1, 2, 3]
b = a[:]
print b
b[0] = 100
print b
print a
Si trabajamos con arreglos de NumPy, el comportamiento es diferente (cuando copiamos, en realidad, estamos haciendo una vista al objeto original, apuntamos al mismo objeto):
a = np.array([1, 2, 3])
b = a[:]
print b
b[0] = 100
print b
print a
¿Cómo resolvemos esta discrepancia?
a = np.array([1, 2, 3])
b = a[:].copy() # fuerzo la copia
print b
b[0] = 100
print b
print a
Escalares:
a = np.array([1, 2, 3, 4])
a + 1
2**a
Aritméticas:
b = np.ones(4) # otra función generadora de arreglos
b = b + 1
b
b - a
a * b
c = np.ones((3, 3))
c
c * c # ojo! esto es una multiplicación elemento por elemento.
c.dot(c) # multiplicación matricial
Lógicas:
a = np.array([1, 2, 3, 4])
b = np.array([4, 2, 2, 4])
a == b
a > b
Otras operaciones lógicas:
a = np.array([1, 1, 0, 0], dtype=bool)
b = np.array([1, 0, 1, 0], dtype=bool)
a | b # or, np.logical_or
a & b # and, np.logical_and
a ^ b # xor, np.logical_xor
not_a = - a # not, np.logical_not
not_a
Calculando sumas:
x = np.array([1, 2, 3, 4])
x.sum() # np.sum(x)
por filas y columnas:
x = np.array([[1, 1], [2, 2]])
x.sum(axis=0) # por columna
x[:,0].sum(), x[:,1].sum()
x.sum(axis=1) # por filas
x[0,:].sum(), x[1,:].sum()
Calculando estadísticos:
x = np.array([1, 2, 3, 1])
x.mean()
x.std()
x.std(ddof=1) # con divisor n - 1
np.median(x)
y = np.array([[1, 2, 3], [5, 6, 1]])
np.median(y, axis=-1) # último eje: en este caso, filas
Encontrando extremos:
x.min()
np.argmin(x) # índice del mínimo
x.max()
np.argmax(x) # índice del máximo
Más operaciones lógicas:
np.all([True, True, False])
np.any([True, True, False])
En NumPy es posible hacer operaciones entre arreglos de diferente tamaño a través del broadcasting.
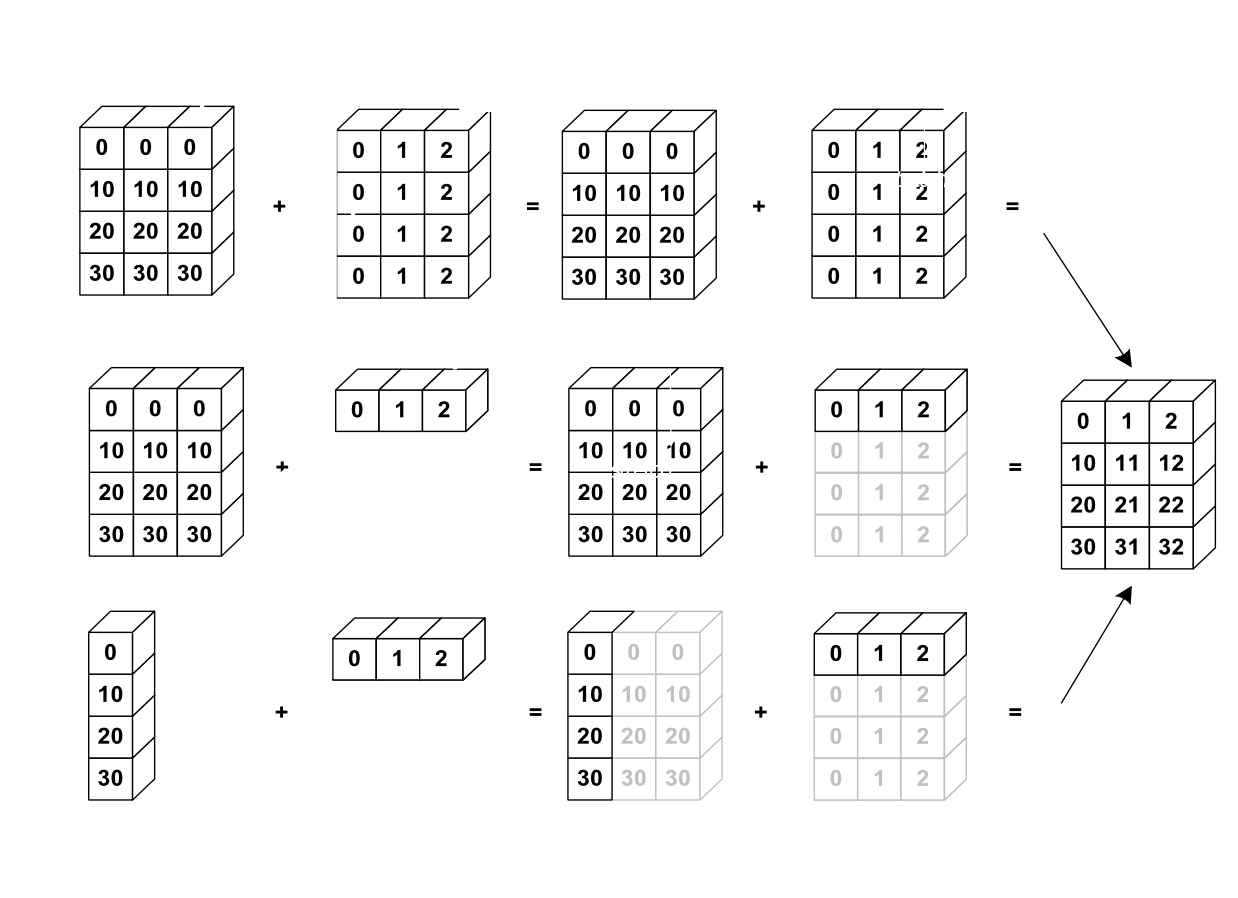
a = np.arange(0, 40, 10)
a.shape
a = a[:, np.newaxis] # suma un nuevo eje (arreglo 2D)
a.shape
a
b = np.array([0, 1, 2])
b
a + b
Multiplicaión de matrices:
a = np.triu(np.ones((3, 3)), 1) # see help(np.triu)
a
b = np.diag([1, 2, 3])
b
a.dot(b)
Trasposición:
a.T
Inversión y resolución de equaciones lineales:
A = a + b
A
B = np.linalg.inv(A)
B
B.dot(A)
x = np.linalg.solve(A, [1, 2, 3])
x
A.dot(x)
Cálculo de autovalores:
np.linalg.eigvals(A)
"Aplanado":
a = np.array([[1, 2, 3], [4, 5, 6]])
a.ravel()
a.T
a.T.ravel()
Cambio de forma:
a.shape
b = a.ravel()
b
b.reshape((2,3))
a = np.arange(36)
b = a.reshape((6,6))
b
b = a.reshape((6, -1)) # -1: comodín, se infiere
b
Cambio de tamaño:
a = np.arange(4)
a.resize((8,))
a
Agregado de nuevas dimensiones:
vector = np.array([1,2,3])
vector
np.shape(vector)
matrix_col = vector[:, np.newaxis] # lo convierto en matrix 3 x 1
matrix_col
np.shape(matrix_col)
matrix_ver = vector[np.newaxis, :] # lo convierto en matrix 1 x 3
matrix_ver
np.shape(matrix_ver)
Repeticiones:
a = np.array([[1, 2], [3, 4]])
a
np.repeat(a, 3)
np.tile(a, 3)
Concatenación:
b = np.array([[5, 6]])
np.concatenate((a, b), axis=0)
np.concatenate((a, b.T), axis=1)
np.vstack((a,b))
np.hstack((a,b.T))
a = np.array([1,0,1,0,0], dtype=bool)
b = np.where(a) # where
b
a = np.array([[1, 2, 3], [4, 5, 6], [7, 8, 9]])
a
np.diag(a) # diag
a = np.arange(10,16)
a
indices = [1, 3, 5]
np.take(a, indices)
which = [0, 0, 1, 0]
choices = [[-2, -3, -4, -5], [5, 6, 7, 8]]
np.choose(which, choices)
x = np.arange(0, 10, 1) # argumentos: inicio, fin, paso
x
np.linspace(0, 10, 25) # incio, fin, n° puntos intermedios
np.logspace(0, 10, 10, base=np.e)
x, y = np.mgrid[0:5, 0:5] # similar al meshgrid de MATLAB
x
y
np.random.rand(5, 5) # números aleatorios uniformes [0,1]
np.random.randn(5,5) # números aleatorios normalmente distribuidos
np.diag([1,2,3]) # matrix diagonal
np.diag([1,2,3], k=1) # desplazamiento sobre la diagonal
np.zeros((3,3))
np.ones((3,3))
Podemos leer archivos csv:
!head stockholm_td_adj.dat
data = np.genfromtxt('stockholm_td_adj.dat')
data.shape
data
pero también podemos escribirlos:
M = np.random.rand(3,3)
M
np.savetxt("random-matrix.csv", M)
!cat random-matrix.csv
np.savetxt("random-matrix.csv", M, fmt='%.5f') # fmt especifica el formato a escribir
!cat random-matrix.csv
np.save("random-matrix.npy", M)
!file random-matrix.npy
np.load("random-matrix.npy")